El Dr. David Romero es investigador del Centro de Ciencias Genómicas de la Universidad Nacional Autónoma de México, en Cuernavaca, Morelos. Su área de especialidad es la genómica bacteriana, con énfasis en mecanismos de cambio en genomas. Es miembro y expresidente de la Academia de Ciencias de Morelos.
Esta publicación fue revisada por el comité editorial de la Academia de Ciencias de Morelos.
¿Qué sería de la vida si no tuviéramos valor para intentar nada?
Vincent van Gogh (1853-1890)
Cuidando el genoma
En el material genético (DNA) de una célula está almacenada la información que le permite realizar sus funciones (Fig. 1). Al conjunto de esa información se le llama genoma y por sí mismo es inerte. Es solo hasta que la información del genoma, contenida en los genes es transcrita a RNA y posteriormente, en la mayoría de los casos, traducida a proteínas, que puede entonces ejercer alguna función biológica. Más aún, todas las células tienen que cuidar que, al momento de multiplicarse para generar nuevas células, estas reciban una copia completa del genoma original. Sí, el genoma debe de copiarse (replicarse es el término que empleamos los biólogos). ¡Imagínense la catástrofe que sería si se generaran nuevas células, pero estas carecieran de un genoma!
Sí, hay que cuidar el genoma. Pero todas las células pueden experimentar adversidades. Muchos agentes naturales, sean estos físicos, químicos o biológicos, pueden dañar al genoma de manera azarosa. Salvo evitar la exposición a los agentes que producen daño al genoma, hay pocas maneras de evitar el daño que puedan causar. Por esa razón, todos los organismos vivos poseen varios mecanismos que reparan el daño causado a los genomas (1). El daño puede ocurrir, pero la efectividad de los mecanismos de reparación es tal que corrigen los peores problemas para un genoma, como partirlo en varios pedazos o perder grandes sectores del genoma. Hacen esto deteniendo el proceso de la división celular, hasta que se corrige el problema. La tarea de los mecanismos de reparación es sorprendente por los tamaños de genoma que tienen que vigilar. Si se trata de bacterias, tienen que monitorear posibles daños en sus genomas, cuyo tamaño oscila entre un millón y diez millones de pares de bases. Si se trata de eucariontes (organismos que encierran su genoma en un núcleo) los tamaños de genoma pueden ser enormes, abarcando desde ocho millones hasta doscientos mil millones de pares de bases, dependiendo del organismo.

Figura 1. El DNA, molécula que constituye el genoma.
Imagen tomada de https://stocksnap.io/photo/dna-helix-CHEZDDXUJY
Pero ningún mecanismo biológico es 100% eficiente, y la reparación del genoma no es la excepción. A una frecuencia baja (de aproximadamente una en cada millón de células) el daño puede no ser corregido, generando una alteración en el genoma, llamada mutación. Esta frecuencia de daño es muy baja y las poblaciones celulares pueden contender con ello. Si la mutación lleva a la inactivación de algún gene esencial para la vida, la célula resultante morirá. La muerte de unas pocas células no constituye un gran problema si hay un número elevado de células en una población. En algunas ocasiones, el cambio provocado por la mutación podría mejorar la función de algún gene, aumentando así la capacidad de supervivencia y/o reproducción de una célula con respecto a otras. De pasar eso, las células que contienen esa mutación tenderán a predominar en la población, por selección natural. Visto de esa manera, la mutación es el motor para la evolución. Todas las células están sujetas a mutación, y esta puede ocurrir cada vez que una célula se divide. En una película de 2002 se decía que “Vivir mata”. Más bien, deberíamos decir que vivir muta.
Construcción de un genoma bacteriano sintético
No es exagerado decir que la generación intencional de mutaciones en genes específicos, así como la caracterización de sus efectos en un organismo, constituyeron los grandes avances de la genética y biología molecular a finales del siglo XX. Estos estudios, acoplados con las facilidades introducidas por la ingeniería genética, permitieron el conocimiento detallado de los genomas. El avance desde luego era lento en esos tiempos, e implicó la participación de un gran número de científicos. Desde entonces, aprendimos que los genomas contienen genes esenciales para las diferentes funciones biológicas básicas (replicación, transcripción, traducción, división celular, etc.) pero que también contienen genes que permiten adaptaciones para ambientes específicos, para resistencia a antibióticos y otras funciones como diferenciación y que estos genes no son usados todo el tiempo, sino que se utilizan solo en circunstancias específicas.
Este conocimiento, junto con avances en métodos para determinar el orden de las bases en grandes segmentos del DNA (secuenciación de DNA) permitieron el nacimiento de las ciencias genómicas a finales del siglo XX. Por primera vez fue posible determinar el orden de todas las bases en un genoma. Al mismo tiempo, se desarrollaron métodos para sintetizar moléculas de DNA con una composición y secuencia de bases conocida y la capacidad para ensamblar estas moléculas sintetizadas químicamente en moléculas cada vez más grandes. Para principios del siglo XXI, comenzó a hablarse de la posibilidad de crear un genoma completo de secuencia conocida por una combinación de síntesis química y ensamble por las técnicas de las ciencias genómicas. La tarea sonaba extremadamente complicada, y fue tachada como de ciencia ficción.
Pero uno de los aspectos fascinantes de la ciencia es que repentinamente lo imposible se vuelve posible. En 2010, el grupo de J. C. Venter-destacado científico corresponsable de la secuenciación de una de las versiones del genoma humano- reportó la síntesis química y ensamble de un genoma bacteriano completo (2). Para ello, este grupo aprovechó el conocimiento de la secuencia genómica completa de la bacteria Mycoplasma mycoides, una bacteria con un genoma relativamente “pequeño” de poco más de un millón de pares de bases. Esta bacteria es innocua para el humano y a pesar de su “pequeño” tamaño de genoma, es capaz de duplicarse con bastante rapidez (en solo una hora) en un medio de cultivo relativamente sencillo.
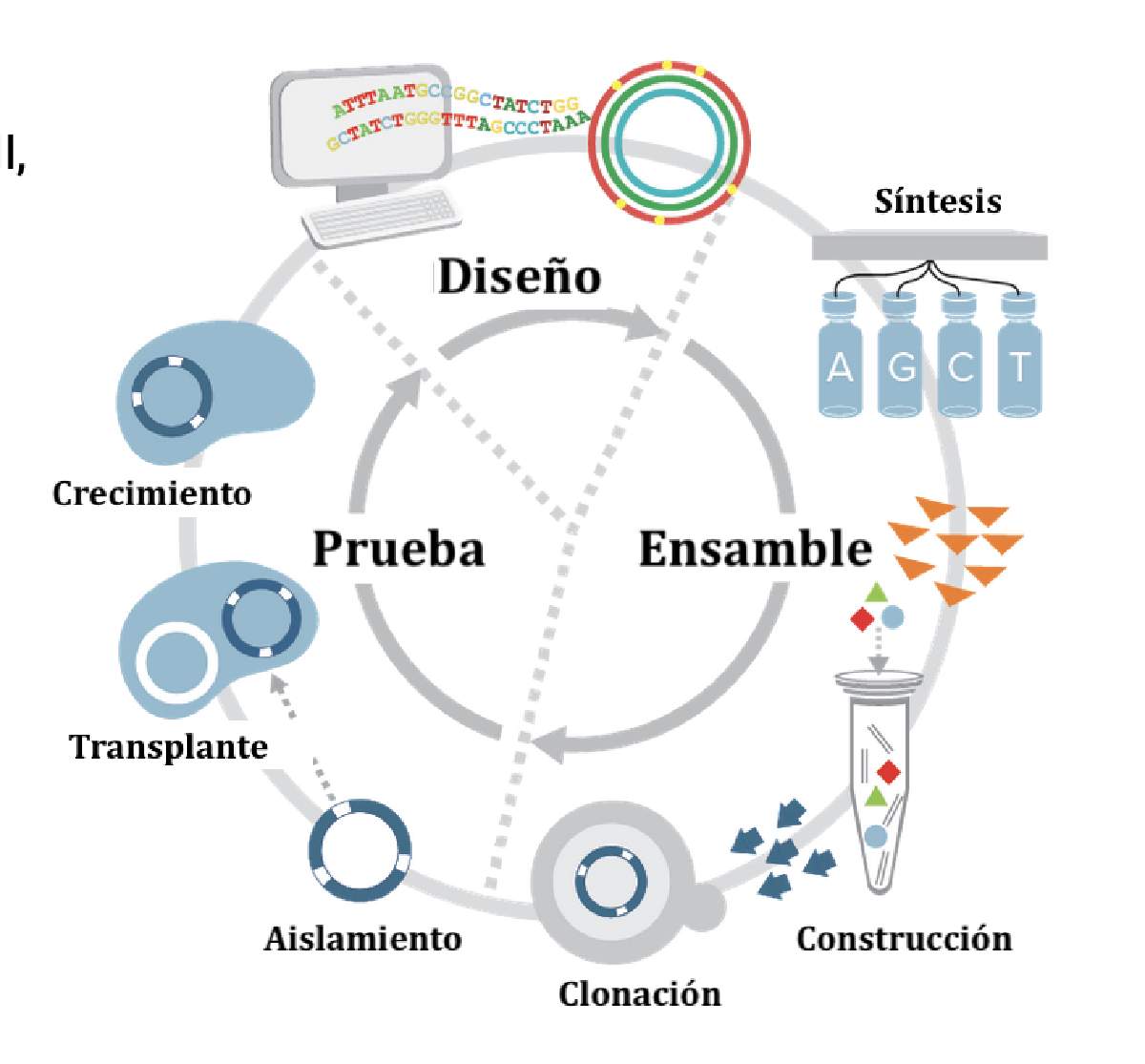
Figura 2. Proceso de generación de células de Mycoplasma mycoides JCVI-syn 1.0, conteniendo un genoma bacteriano sintético. Imagen adaptada de una tomada de https://www.jcvi.org/research/first-minimal-synthetic-bacterial-cell
El proceso que siguieron para la generación de un genoma bacteriano sintético se muestra en la Fig. 2. Para ello, hicieron un diseño asistido por computadora de los múltiples segmentos a sintetizar, cada uno de tamaño pequeño. Durante este diseño, introdujeron varios cambios en la secuencia para usarse como “marcas” del origen sintético de cada secuencia. Cada segmento se sintetizó químicamente y se fueron ensamblando estos segmentos en un orden predeterminado que correspondía al de M. mycoides original. Este proceso se repitió hasta lograr el ensamble de un genoma circular completo para esta bacteria. El siguiente paso, consistió en el “transplante” de este genoma sintético a otra bacteria. Para ello, introdujeron el genoma sintético de M. mycoides en células de otra bacteria relacionada, Mycoplasma capricolum. Lo que se esperaba es que el genoma sintético desplazara por completo al genoma de M. capricolum, dejando una nueva célula conteniendo únicamente al genoma sintético. Lo esperado funcionó, llevando a la generación del primer organismo conteniendo un genoma bacteriano completamente sintético, llamado Mycoplasma mycoides JCVI-Syn1.0. Este nuevo organismo, a pesar de portar un genoma sintético, posee las mismas características biológicas, incluyendo velocidad de crecimiento, que Mycoplasma mycoides.
Reduciendo un genoma a lo indispensable
El éxito en generar M. mycoides JCVI-Syn1.0 fue un logro notable, demostrando que era posible generar una célula en el cual su genoma original fue sustituido por uno rediseñado y construido en el laboratorio. Pero hay que admitir que, a pesar de los cambios introducidos intencionalmente durante el diseño, este genoma seguía siendo prácticamente idéntico al genoma original de M. mycoides. El siguiente reto era más difícil de lograr. ¿Será posible generar un genoma sintético que contenga sólo los genes necesarios, esenciales para supervivencia y reproducción? Sí, un genoma reducido, minimizado con respecto al original. Para ese entonces se contaba con la secuencia de otros genomas de bacterias con lo que se realizó una comparación, con la finalidad de saber qué genes están conservados en diferentes organismos y por ende, asumir que dichos genes deben ser esenciales para la vida. Estos análisis y otros más de tipo experimental permitieron la localización en el genoma de M. mycoides de los genes esenciales para este organismo.
Desde luego, estos genes no estaban contiguos en el genoma, sino que estaban dispersos. Si se deseaba sintetizar un genoma minimizado, esto significaba que debería diseñarse de manera que permitiera unir segmentos alejados en el genoma, excluyendo aquellos segmentos que contenían genes no esenciales. Un problema que podría presentarse es que nadie tenía la certeza de que se hubieran identificado todos los genes esenciales en ese genoma. Bastaría la exclusión de solo un gene esencial para que la generación de un genoma minimizado fallara.
El diseño y síntesis del genoma minimizado se hizo de manera similar al mostrado en la Fig. 2, esperando generar un genoma minimizado. Esta versión minimizada contendría menos de la mitad del genoma original de esta bacteria. El esfuerzo se hizo… y fallaron. Fue imposible obtener una célula viva que contuviera solo esta versión reducida. Evidentemente algo esencial para la vida estaba faltando en este genoma minimizado. El identificar cuál o cuáles genes estaban faltando requirió un enorme esfuerzo adicional de experimentación, pero este esfuerzo rindió frutos. En 2016 se logró generar células vivas conteniendo un genoma minimizado, llamadas Mycoplasma mycoides JCVI-Syn3.0 (Fig. 3), conteniendo solo la mitad del genoma original (531 mil pares de bases, en vez de un millón setenta y ocho mil pares de bases) y poco más de la mitad de los genes originales (473 en lugar de 911).
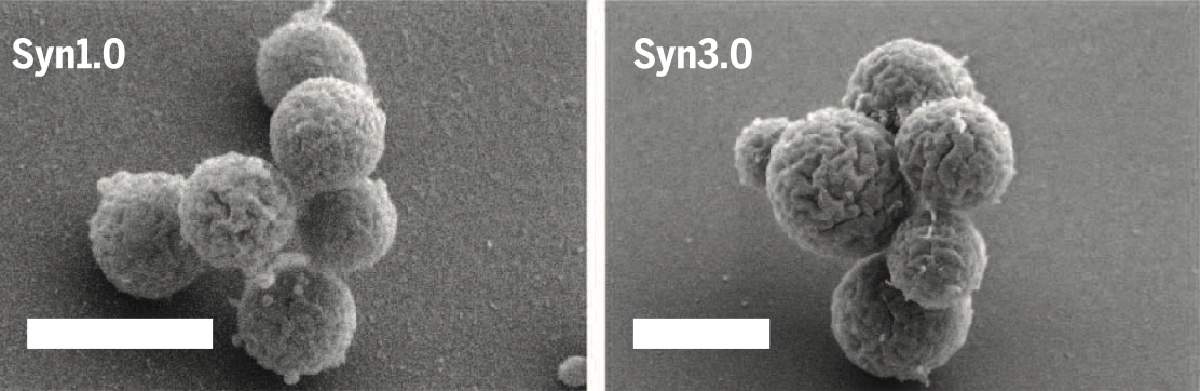
Figura 3. Células de Mycoplasma mycoides conteniendo un genoma idéntico al original (Syn1.0) o un genoma minimizado (Syn3.0). Imagen tomada de la referencia 3.
Por primera vez se contaba con células conteniendo un genoma minimizado, diseñado en el laboratorio, conteniendo solo los genes esenciales para la vida. Esto atrajo un gran interés. El contar con células que contienen solo lo esencial para la vida abre posibilidades prácticas para generar, por técnicas de biotecnología moderna, células que empleen sus recursos para producir proteínas de interés. Desde un punto de vista básico, el contener solo un genoma minimizado nos acerca a una situación similar a la experimentada hace miles de millones de años en las primeras células que habitaron nuestro planeta.
Pero estas células minimizadas enfrentan también la adversidad. Si bien las células con un genoma minimizado se ven similares a las células originales, son más pequeñas, más variables en tamaño y se dividen más lentamente (3 h en lugar de sólo 1 h en las células originales) además de que solo pueden subsistir en un medio de cultivo especial en condiciones de laboratorio. Estas características hacen improbable que puedan sobrevivir en condiciones más naturales.
Superando la adversidad en un genoma minimizado
¿Cómo superar estos efectos adversos en crecimiento? El grupo de J. C. Venter abordó este problema generando otra versión de sintética de M. mycoides, Syn 2.0, que contenía algunos genes adicionales y tenía un tiempo de división de 1.5 h. Interesante, sí, pero en cierto sentido era obvio que al añadir más genes de los originales iba a mejorar el crecimiento. Otros grupos razonaron el cómo superar esta adversidad con un enfoque basado en evolución. Los grupos de J. T. Lennon (4) y B. O. Palsson (5), trabajando en E.E.U.U., razonaron que si se cultivaba continuamente en el laboratorio a Syn3.0 podrían aparecer mutaciones en su genoma que mejoraran la capacidad de duplicación de las células. Sí, ambos grupos se propusieron hacer evolución en el laboratorio. Desde luego, era imposible saber cuánto tiempo deberían de cultivarse esas células para encontrar mutantes con un crecimiento mejorado, o si era posible siquiera. Existen otros ejemplos de evolución en el laboratorio, pero estos se habían hecho siempre con organismos naturales, seleccionando para una propiedad, pero nunca empleando un genoma minimizado.
Ambos grupos publicaron sus resultados hace poco menos de un año. El grupo de Palsson cultivó continuamente M. mycoides, Syn 3.0 por un tiempo equivalente a 800 generaciones. Al final de este periodo, las células habían mejorado su capacidad de duplicación en un 15% (5). El grupo de Lennon fue más persistente, cultivando estas células por 2000 generaciones (aproximadamente 300 días), encontrando que estas células habían mejorado su crecimiento, dividiéndose ahora solamente en una hora, en lugar de las 3 h iniciales (4). Paralelamente, el grupo de Lennon cultivó también M. mycoides, Syn 1.0 las mismas condiciones, encontrando que aún en estas células puede mejorarse su tiempo de duplicación, pero solo en un 50%. Las células de Syn3.0 (genoma minimizado) parecen adaptar su crecimiento con mayor rapidez que las células de Syn1.0.
Ambos grupos secuenciaron completamente los genomas de las células adaptadas, buscando qué genes habían tenido cambios que permitieran mejorar el crecimiento. Ambos grupos encuentran mutaciones en genes que participan en la síntesis de lípidos, componentes necesarios en la membrana que rodea las células. El grupo de Lennon encuentra también mutaciones en otro gene, ftsZ, que participa en división celular. Pero estos no son los únicos cambios, se encontraron mutaciones en 15 genes adicionales, cuyos efectos exactos están aún por aclararse.
Estos resultados son muy interesantes. Muestran claramente que los efectos adversos en el crecimiento pueden superarse a través de selección para un mejor crecimiento, en un tiempo bastante rápido. Es de esperarse que estos estudios continúen, para aclarar el papel de las diferentes mutaciones para lograr un mejor crecimiento. Pero es probable también que experimentos futuros de evolución en el laboratorio añadan nuevos elementos. En los experimentos actuales, cada célula en la población puede o no experimentar mutaciones que mejoren, aún en pequeña magnitud, su capacidad de crecimiento. Si lo logran, aumentarán su representación en la población. Mejorías adicionales requieren que cada célula en lo individual adquiera espontáneamente nuevas mutaciones. Pero la evolución tiene un mecanismo adicional para compartir mejorías entre células. Este mecanismo, conocido como transferencia genética horizontal, es capaz de generar nuevas combinaciones al transferir nuevas mutaciones de una célula a otra. Es un mecanismo capaz de producir nuevas combinaciones de mutaciones dentro de una población. Syn3.0 carece de este mecanismo. ¿Podrá mejorarse aún más y más rápido la capacidad de adaptación al introducir la posibilidad de transferencia horizontal? La idea es al menos razonable. Es un momento emocionante para dedicarse a la investigación científica, particularmente en las ciencias genómicas. Recordando a van Gogh, ¿Qué sería de la vida si no tuviéramos valor para intentar nada?
Referencias
1) Reparación del ADN https://es.wikipedia.org/wiki/Reparaci%C3%B3n_del_ADN
2) Gibson, D. G., Glass, J. I., Lartigue, C., Noskov, V. N., Chuang, R.-Y., Algire, M. A., et al. (2010). Creation of a Bacterial Cell Controlled by a Chemically Synthesized Genome. Science 329, 52–56. doi: 10.1126/science.1190719
3) Hutchison, C. A., Chuang, R.-Y. Y., Noskov, V. N., Assad-Garcia, N., Deerinck, T. J., Ellisman, M. H., et al. (2016). Design and synthesis of a minimal bacterial genome. Science (New York, N.Y.) 351, aad6253. doi: 10.1126/science.aad6253
4) Moger-Reischer, R. Z., Glass, J. I., Wise, K. S., Sun, L., Bittencourt, D. M. C., Lehmkuhl, B. K., et al. (2023). Evolution of a minimal cell. Nature 620, 122–127. doi: 10.1038/s41586-023-06288-x
5) Sandberg, T. E., Wise, K. S., Dalldorf, C., Szubin, R., Feist, A. M., Glass, J. I., et al. (2023). Adaptive evolution of a minimal organism with a synthetic genome. iScience 26, 107500. doi: 10.1016/j.isci.2023.107500
Esta columna se prepara y edita semana con semana, en conjunto con investigadores morelenses convencidos del valor del conocimiento científico para el desarrollo social y económico de Morelos. Desde la Academia de Ciencias de Morelos externamos nuestra preocupación por el vacío que genera la extinción de la Secretaría de Innovación, Ciencia y Tecnología dentro del ecosistema de innovación estatal que se debilita sin la participación del Gobierno del Estado.


